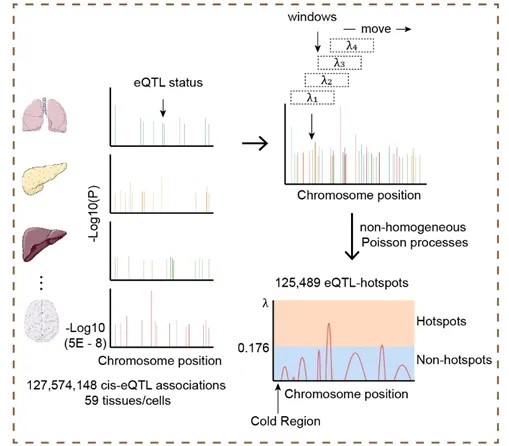
近年来,表达数量性状位点(eQTL)研究蓬勃发展,为我们深入理解真核细胞的转录调控机制以及全基因组关联研究(GWAS)中遗传变异影响的生物学过程提供了重要线索。当前,大多数eQTL研究都采用严格校正的P值,以确保鉴定高度可信的eQTL位点。然而,严格控制错误发现率也限制了识别真实eQTL的效力。因此,厦门大学医学院统计支持办公室近期上线了一种表观基因组增强的eQTL分析框架,E-SpotFinder-XMBD[1],基于泛组织eQTL热点建模,预测组织特异性的eQTL热点,此项工作大大了提高识别真实eQTL位点的能力。 一、E-SpotFinder-XMBD框架原理和优势 我们首先提出了一种基于非齐次泊松过程的方法,在59种人类组织或细胞类型中识别出了125,489个eQTL高频出现的区域(eQTL热点)。
我们根据不同的序列特征将eQTL热点分为两类:C1和C2。这两类eQTL热点均在活跃的表观遗传学标志物(例如H3K27ac、H3K4me1和H3K4me3)中呈现高活性,在抑制性表观遗传学标志物(例如H3K9me3)中呈现低活性。eQTL热点C1主要参与非活性启动子相关的活动,eQTL热点C2主要参与转录起始和增强子相关的活动。其次,我们基于这两类eQTL热点及其表观基因组学和基因组学特征,建立了名为“E-SpotFinder-XMBD”的机器学习模型,用于增强发现组织或细胞类型特异性eQTL热点。
二、性能对比 我们将此模型应用于36种组织或细胞类型中,发现组织或细胞类型特异的eQTL热点与泛组织热点具有相同的调控特性,且展现出与增强子相似的高水平eQTL 活性。最后,我们在36种组织或细胞类型的eQTL热点内重新定义了eQTL显著性阈值。与其他阈值方法相比,由此得到的eQTL在精准定位的eQTL和疾病的风险位点中呈现出更高的富集倍数。
综上所述,我们建立了一个表观遗传学增强的eQTL分析框架,可预测各组织或细胞类型中的eQTL热点即DNA顺式调控热点,这有助于更好地理解基因的转录调控。 运行E-SpotFinder 1. python E-SpotFinder.py <input_feature_matrix> 例子 1. git clone git@github.com:Lhhuan/E-SpotFinder.git 2. 3. cd E-SpotFinder 4. 5. python E-SpotFinder.py Example_input.txt.gz 依赖模块 1. Python: 3.9.12 2. 3. pandas: 1.2.5 4. 5. numpy: 1.20.3 6. 7. pickle: 4.0 8. 9. xgboost: 1.5.0 E-SpotFinder的引用信息请参考: LIU H, CHEN Q, GUO J, et al. Epigenome-augmented eQTL-hotspots reveal genome-wide transcriptional programs in 36 human tissues[J]. Brief Bioinform, 2024, 25(3):bbae109. 三、厦门大学医学院统计支持办公室新实验方法咨询 厦门大学医学院统计支持办公室提供基于E-SpotFinder-XMBD框架的定制化组织特异性的DNA顺式调控热点分析流程,主要服务内容包括: (1)数据分析:包含实验数据整理、统计分析、高通量测序分析、图表设计及绘制和相关解释。 (2)咨询服务:提供统计方法、图表设计、样本量计算咨询。 (3)分析报告:包括数据分析结果总结和科研论文方法部分的撰写。 我们十分乐意为老师和同学们解答E-SpotFinder分析过程中的各种问题。无论是在数据处理、分析方法选择还是科研论文撰写阶段,我们都愿意为同学们提供专业的建议和支持! 地点:医学院健康医疗大数据国家研究院爱礼楼201室 联系电话:18030257072 邮箱:trans_medxmu@126.com 参考文献: [1] LIU H, CHEN Q, GUO J, et al. Epigenome-augmented eQTL-hotspots reveal genome-wide transcriptional programs in 36 human tissues[J]. Brief Bioinform, 2024, 25(3): bbae109 |