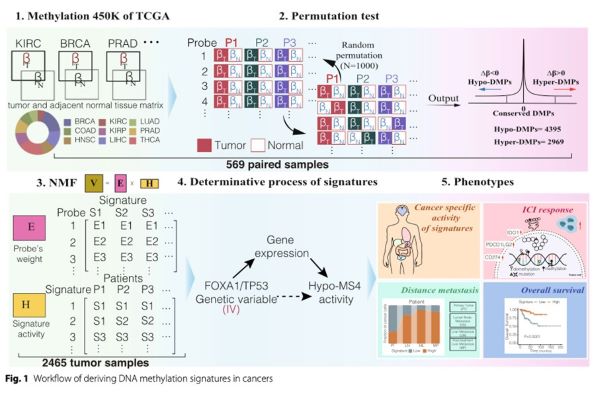
近日,厦门大学医学院统计支持办公室近期在肿瘤表观遗传学研究方面取得重要进展, 研究团队开发了一种基于置换检验和非负矩阵分解的整合泛癌分析DNA甲基化特征识别方法(PROMISEXMBD, PeRmutation-based methOlation sIgnature identification using nonnegative Matrix factorization from Integrative pan-cancer analySEs)。研究团队利用了大规模的置换检验的统计学方法(permutation test)和非负矩阵分解(NMF) 的方法,从TCGA的2,465个癌症甲基化谱和基因组药物敏感性癌症(GDSC)队列的241个细胞系甲基化谱中提取了独立的甲基化特征, 并将DNA甲基化特征与各种临床和生物学特征相关联,包括年龄、生存率、癌症分期、肿瘤免疫微环境因素和免疫治疗反应。其研究成果发表在《Genome Medicine》。
PROMISEXMBD方法亮点1. 大规模的置换检验, large scale Permutation test, 用于鉴定差异甲基化位点,通过对569对癌症组织和匹配正常组织的DNA甲基化谱进行配对排列,定义了7364个差异甲基化位点(2969个高甲基化DMPs和4395个低甲基化DMPs)。 2. 非负矩阵分解,用于从多个癌症数据集中提取独立的甲基化特征(signature identificantion),从癌症组织和细胞系中提取了三个高度保守的独立甲基化特征(Hyper-MS1、Hypo-MS1和Hypo-MS4)。 3. 整合泛癌分析, Integrative pan-cancer analyse, ,即整合TCGA和GDSC等多个癌症数据集进行分析。 4.临床和生物学特征关联:将DNA甲基化特征与年龄、生存率、癌症阶段、肿瘤免疫微环境因素和免疫治疗反应等临床和生物学特征相关联。 5. 基因组和转录组数据整合:通过整合基因组和转录组数据,推断这些甲基化特征的决定基因。
自助分析流程研究者可以在R环境下方便地构建自己的甲基化特征分析流程:
1. R包安装 install.packages("predictmeans") install.packages("NMF") install.packages("stringr") install.packagrs("foreach") install.packages("doParallel") 2. 快速开始 置换检验的运行 #数据准备 data_meth <- read.csv("450K.csv") ##读入甲基化芯片数据 clinilcal_info <- read.csv("sampels_info.csv") ##读入样本临床信 #运行置换检验 m1 <- lmer(betaval ~ status * cancertype + (1|TSS), control=lmerControl(check.conv.singular = .makeCC(action = "ignore", tol = 1e-8))) ###多元回归分析 perm <- permanova.lmer(m1,perms = 1E3) ###置换检验运行 result <- data.frame(cg = i,status_p = perm$Perm.p[1], cancertype_p = perm$Perm.p[2], status_cancertype_p = perm$Perm.p[3]) ##提取置换检验结果 甲基化标签提取 #数据准备 perm_result <- read.csv("permutation_result_probe_450K.csv") ##读入筛选的甲基化芯片矩阵切不含缺失值;行:探针,列:样本名, #NMF的标签提取 nmf_run <- nmf(da,2:8,'ns',seed = 123456,nrun=100) nmf_run_ns <- nmf_run$fit[[k]] ##k是目标选择的标签数 signature <- predict(nmf, what= "features",prob = TRUE) ##获得甲基化标签 其他分析流程参照https://github.com/xmbd/Pan_Meth_Sig 研究成果亮点该研究揭示了DNA甲基化特征在肿瘤免疫微环境塑造和免疫治疗反应预测中的重要作用,为癌症精准医疗提供了新的思路。相关研究成果已发表在Genome Medicine,2024。 PROMISEXMBD方法的引用信息请参考:1. Qin Q, Zhou Y, Guo J, et al. Conserved methylation signatures associate with the tumor immune microenvironment and immunotherapy response [J]. Genome Med, 2024, 16(1): 47.
厦门大学医学院统计支持办公室新实验方法咨询 厦门大学医学院统计支持办公室提供基于PROMISEXMBD框架的定制化甲基化分析流程,主要服务内容包括: (1)数据分析:包含实验数据整理、统计分析、高通量测序分析、图表设计及绘制和相关解释。 (2)咨询服务:提供统计方法、图表设计、样本量计算咨询。 (3)分析报告:包括数据分析结果总结和科研论文方法部分的撰写。 我们十分乐意为老师和同学们解答PROMISEXMBD分析过程中的各种问题。无论是在数据处理、分析方法选择还是科研论文撰写阶段,我们都愿意为同学们提供专业的建议和支持! 地点:医学院健康医疗大数据国家研究院,爱礼楼201 联系电话:18030257072 邮箱:trans_medxmu@126.com
参考文献: 1. Qin Q, Zhou Y, Guo J, et al. Conserved methylation signatures associate with the tumor immune microenvironment and immunotherapy response [J]. Genome Med, 2024, 16(1): 47. 2. Dongwen Luo, Siva Ganesh and John Koolaard, https://CRAN.R-project.org/package=predictmeans. 3. Gaujoux R, Seoighe C. A flexible R package for nonnegative matrix factorization [J]. BMC Bioinformatics, 2010, 11: 367.
|